Matlab中分层聚类
一般分层聚类分为以下几步:
分步聚类:(1)用pdist函数计算样本之间的距离,确定两两样本之间的距离或相似性(这个和选择的计算pdist的方法有关系);(2)用linkage函数定义之间的连接;(3)用cophenet函数评价聚类效果;(4)用cluster函数进行聚类。
clusterdata 一个可完整实现聚类算法的函数,是以下pdist、linkage和cluster函数的综合。
pdist 计算样本点之间的距离,一般在计算距离之前用zscore函数对数据做标准化;
(另外,可以用squareform对pdist得到的距离用距离矩阵的形式呈现)
% Y = pdist(X);
linkage 以pdist的距离为输入,根据两两对象之间的距离进行聚类,输出为两两节点连接的顺序和他们之间的距离;
% Z = linkage(Y);
dendrogram 绘制linkage得到的聚类树,倒U型的高度代表对象间的距离
% dendrogram(Z);
cophenet 验证距离树的合理性,即对比cohpenetic距离与pdist函数计算的原始数据的距离之间的相关性,当然,相关值越接近于1说明聚类效果越好
% c = cophenet(Z, Y)
inconsistent 列出聚类树中各连接的不一致系数,这个值通过比较连接的高度和其下层连接的平均高度来计算,因此对于两个明显区别聚类的连接,其不一致系数较高。
% I = inconsistent(Z)
cluster 设定阈限(不一致性次数的阈值或最大cluster的个数),对原始数据进行聚类
% T = cluster(Z, ‘cutoff’, 1)
下面是一个例子:
采用猫的脑区连接信息。
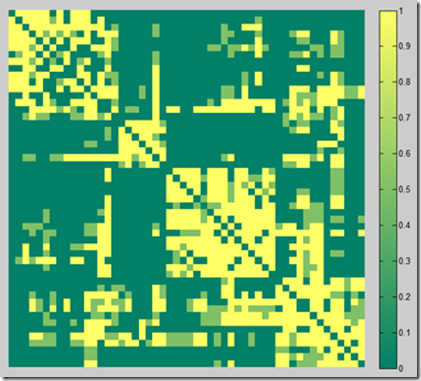
根据连接pattern对脑区进行聚类,代码如下:
catmatfile = ‘./toolbox/BCT/cat.mat’;
catmat = load(catmatfile);
X = (catmat.CIJctx’ + catmat.CIJctx)/2; % 只是为了把原始的连接矩阵搞成对称的
Y = pdist(X, ‘cityblock’);
Z = linkage(Y);
c = cophenet(Z, Y);
% c = 0.7254 in this analysis
dendrogram(Z, ‘LABELS’, catmat.Names);
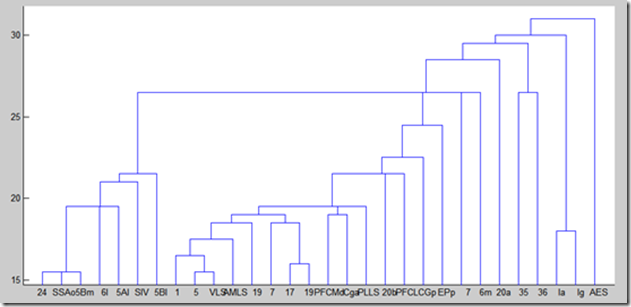
根据上面计算cophenetic系数为0.7254,结果不是很烂;
从聚类树上看,某些脑区可以根据其相似的连接pattern聚为相应的几类。
- Blog Link: http://conxz.net/2013/10/12/matlab-hierarchical-clustering/
- Copyright Declaration: The author owns the copyright (CC BY-NC-ND 4.0).